David Baker团队开发新型AI蛋白设计模型——LigandMPNN,实现原子上下文条件蛋白序列设计
来源:生物世界 2025-04-01 11:31
研究团队开发了一种新型深度学习方法——LigandMPNN,该方法明确地对生物分子系统中的所有非蛋白质成分进行了建模。
蛋白质是生命活动的核心执行者,而通过计算设计新型蛋白质(例如酶、药物结合蛋白)是蛋白质设计领域的“圣杯”。传统设计方法(例如Rosetta)依赖物理模型,耗时且无法精准处理蛋白质与金属离子、小分子等非蛋白成分的相互作用。而目前最先进的基于深度学习的蛋白质序列设计方法(例如ProteinMPNN)虽高效,却“看不见”这些关键元素,无法对其建模,限制了其在药物设计等场景的应用。
那么,如何让 AI 既懂蛋白质结构,又能感知周围化学环境呢?
2025 年 3 月 28 日,诺奖得主、蛋白质设计先驱 David Baker 教授在 Nature Methods 期刊发表了题为:Atomic context-conditioned protein sequence design using LigandMPNN 的研究论文。
该研究开发了一种新型深度学习方法——LigandMPNN,该方法明确地对生物分子系统中的所有非蛋白质成分进行了建模,预计 LigandMPNN 将在设计新的结合蛋白、传感器和酶方面得到广泛应用。

蛋白质的从头设计,能够创造出具有新功能的新型蛋白质,例如催化作用、与 DNA、小分子和金属的结合以及蛋白质间的相互作用。
从头设计通常分三步进行:第一步,生成预测为执行新所需功能接近最优的蛋白质骨架;第二部,为每个骨架设计氨基酸序列,以驱动折叠成目标结构,并形成实现功能所需的特定相互作用(例如,酶活性位点);第三部,使用结构预测方法进行序列 - 结构兼容性筛选。
对于其中关键的第二部,即蛋白质序列设计,可以通过基于物理的方法(例如 Rosetta)以及基于深度学习的模型(例如 ProteinMPNN、IF-ESM 等)来进行。基于深度学习的方法在设计蛋白质主链序列方面优于基于物理的方法,但目前可用的深度学习模型均无法纳入非蛋白的原子和分子。例如,ProteinMPNN 明确只考虑蛋白质主链的坐标,而忽略任何其他原子环境,这导致其在设计酶、核酸结合蛋白、传感器以及所有涉及与非蛋白原子相互作用的其他蛋白质功能时面临困难。
为了实现上述广泛的蛋白质功能的设计,研究团队开发了一种新型深度学习方法——LigandMPNN,该方法明确地对生物分子系统中的所有非蛋白质成分进行了建模。
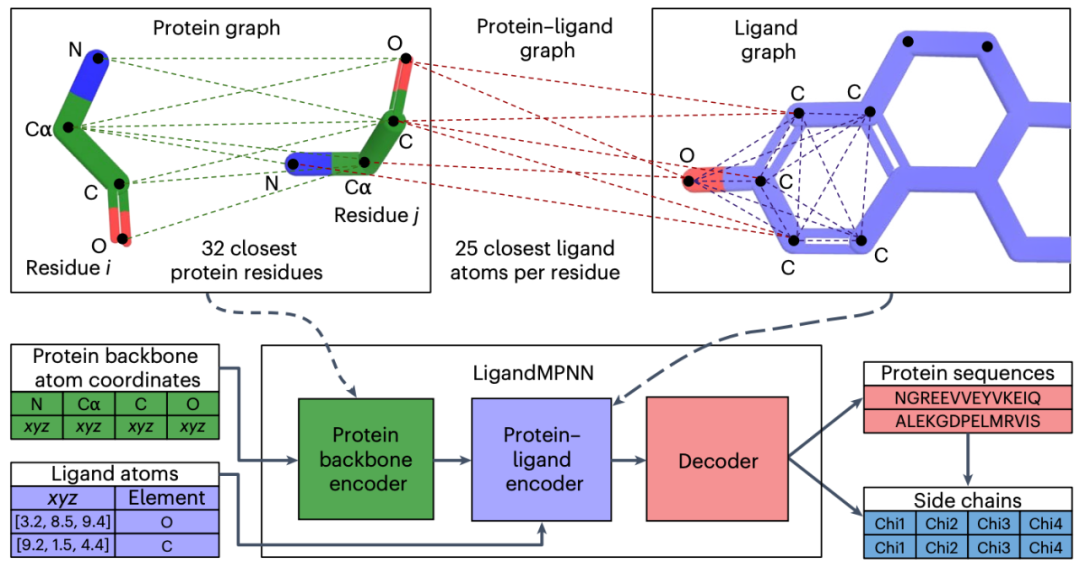
LigandMPNN 的三大创新
1、全局感知的分子图谱
蛋白质-配体交互网络:将蛋白质残基与配体原子(小分子、金属等)构建为图结构,通过距离和化学元素编码相互作用,模拟真实生物环境。
动态信息传递:引入两层神经网络,分别在配体内部原子间、蛋白质与配体间传递信息,捕捉氢键、疏水作用等关键细节。
2、高效侧链建模
一步到位设计:传统方法需分步优化序列和构象,而 LigandMPNN 同步预测氨基酸序列及侧链扭转角,生成可直接评估结合力的 3D 模型。
混合分布预测:采用环形正态分布模拟侧链自由度,提升组氨酸(金属结合的关键)等残基的构象准确性。
3、数据增强与泛化能力
侧链原子模拟配体:在训练中随机将 2%-4% 的蛋白质侧链视为“假配体”,增强模型对结合位点的敏感度。
噪声抗干扰训练:对输入坐标添加高斯噪声(0.1Å),防止模型死记硬背晶体结构,提升对新骨架的适应力。
性能碾压:用实验数据说话
在与小分子、金属以及核苷酸相互作用的氨基酸残基的天然主链序列恢复方面,LigandMPNN 全面优于 Rosetta 和 ProteinMPNN:
与小分子相互作用:LigandMPNN(63.3%)vs. Rosetta(50.4%)vs. ProteinMPNN(50.5%);
与金属离子相互作用:LigandMPNN(77.5%) vs. Rosetta(36.0%)vs. ProteinMPNN(40.6%);
与核苷酸相互作用:LigandMPNN(50.5%) vs. Rosetta(35.2%)vs. ProteinMPNN(34.0%);
此外,LigandMPNN 不仅生成主链序列,还能更精准地生成侧链构象,从而能够对结合相互作用进行详细评估。
实验验证成功案例:
LigandMPNN 已被用于设计超过 100 种经实验验证的小分子和 DNA 结合蛋白,这些蛋白具有高亲和力和高结构准确性(由四个 X 射线晶体结构所表明),并且对 Rosetta 小分子结合剂设计的重新设计使结合亲和力提高了多达 100 倍。
应用前景
药物开发:设计高亲和力抗体或酶,加速靶向疗法。
生物传感器:定制结合金属/毒素的蛋白,用于环境监测。
合成生物学:构建人工代谢通路中的关键酶元件。
值得一提的是,研究团队已在 GitHub 上开源了 LigandMPNN 代码,链接:https://github.com/dauparas/LigandMPNN 。
总的来说,LigandMPNN 不仅是一次技术迭代,更是蛋白质设计范式的革新。当 AI 开始“看见”生命的化学细节,我们距离定制化生物解决方案的时代又近了一步。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


